
| Confectii | Diverse | Film televiziune | Fotografie | Pescuit |
Pescuit
|
|
Qdidactic » dezvoltare & ... » pescuit Construirea arborilor filogenetici |
Construirea arborilor filogenetici
Construirea arborilor filogenetici
Estimarea relatiilor filogenetice pornind de la informatia continuta in secventele ADN se bazeaza pe definirea acelei succesiuni de etape (algoritmi) prin care se poate construi cel mai bun arbore filogenetic. Un arbore filogenetic constituie reprezentarea grafica a filogeniei unui grup de oragnisme. Pentru obtinerea arboilor filogenetici se respecta urmatoarea succesiune de etape:
obtinerea secventelor ADN pentru analiza;
compararea secventelor obtinute cu cele din bancile de date;
alinierea secventelor;
selectarea metodelor filogenetice;
construirea arborilor si evaluarea acestor (Figura 13).
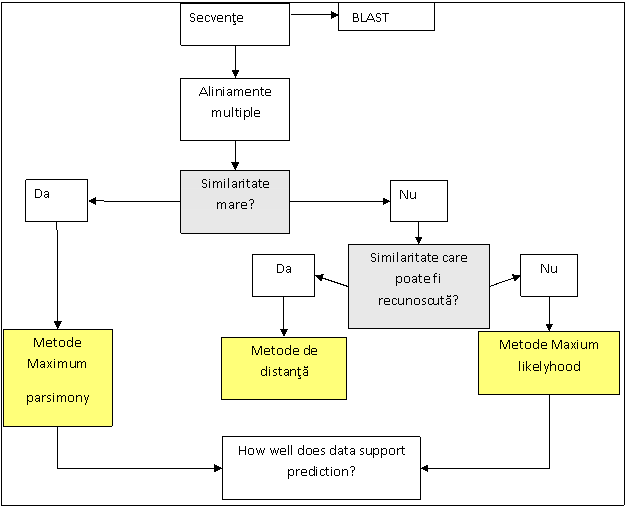
Figura 13. Etapele unei analize filogenetice (D.W.Mount, Bioinformatics: Sequence and Genome Analysis, Cold Spring Harbor Press, 2004)
Alinierea secventelor
Baza fundamentala a unui arbore filogenetic este realizarea unui aliniament multiplu continand secventele de interes. Prin alinierea secventelor se confirma ca toate secventele sunt omologe. Alnierea secventelor se poate realiza manual (folosind un editor text), automat (cu ajutorul unui software specializat) sau combinat.
Secventele obtinute pentru ARNr 16S, ARNr 12S si ARNt la speciile de salmonide analizate au fost aliniate cu programul ClustalX. Pentru alinierea propriu-zisa a secventelor de interes cu programul ClustalX se realizeaza pasii urmatori:
1. se aleg secventele care au acelasi sens de citire (forward sau reverse),
2. secventele sunt trecute fie individual, fie impreuna intr-un format care sa fie recunoscut de programul de aliniere. ClustalX recunoaste datele in format FASTA.
3. se definesc parametrii pentru aliniere (gap penalty). O pereche de secvente poate fi aliniata astfel incat sa maximizam numarul de resturi nucleotidice care sunt comune, prin introducerea de spatii (gap) la una dintre secvente. Biologic, aceste gap se presupun a reprezenta insertii sau deletii care se produc atunci cand secventele sunt divergente dintr-un ancestor comun. Daca inseram prea multe spatii aliniamentul poate deveni fara inteles, este necesar sa restrangem numarul de spatii introduse astfel incat aliniamentul sa aiba sens din punct de vedere biologic. De aceea, este utilizat un sistem de notare astfel incat resturile nucleotidice verificate sa obtina un scor pozitiv si spatiile (gap) sa obtina un scor negativ sau gap penalty (penalizarea discontinuitatilor).
ClustalX creaza aliniamente multiple in trei etape: i) aliniaza individual fiecare secventa cu celellalte secvente intr-o serie de aliniamente perechi; ii) utilizeaza acest set de aliniamente perechi pentru a crea un arbore ghid; iii) utilizeaza acest arbore ghid pentru a crea aliniament multiplu.
Alegerea metodelor de constructie arborilor
In general se utilizeaza doua categorii de metode pentru construirea arborilor filogenetici:
metode de distanta (fenetice).
metode de caractere (cladistice).
Metodele de distanta (fenetice) propun construirea arborilor plecand de la asemanarile observate intre fiecare pereche de unitati evolutive si se bazeaza pe calculul unei matrice de distanta obtinuta comparand secventele doua cate doua si calculand numarul total de diferente pentru toate cuplurile posibile. Se construieste apoi un arbore filogenetic ale carui ramuri sunt cat mai apropiate de numerele continute de matricea de distanta. Cele mai utilizate metode de distanta sunt reprezentate de NJ (Neighbor Joing method), UPGMA (Unweighted Pair-Group Method with Arithmetic means) si presupun construirea arborelui filogenetic prin succesiunea de pasi urmatoare: aliniere→matrice de distanta→arbore filogenetic.
|
Cea mai simpla metoda de a masura distanta dintre doua secvente este de a alinia secventele si de a numara numarul de diferente dintre acestea. Gradul de divergenta se numeste distanta Hamming (d). Pentru un aliniament cu lungimea N si n situsuri, la nivelul caruia exista diferente d = n/N. Distanta observata (D) dintre doua secvente este: D = 1 - S, unde S = M/L. Similitudinea (S) dintre doua secvente reprezinta raportul dintre numarul de situsuri sinonime (M ) si lungimea secventei (L). Distanta evolutiva dintre doua secvente este egala cu numarul de substitutii care sunt produse pe doua linii evolutive plecand de la un ancestor comun. Diferentele observate nu sunt egale cu distanta genetica, aceasta implicand mutatii care nu pot fi observate direct. Convergenta, reversia si schimbarile multiple sunt cel mai adesea ascunse observatiei. Pentru a tine cont de ele, numeroase modele au fost dezvoltate pentru a corecta distantele observate. Jukes si Cantor (1969) au propus o formula corectiva: D = -3/4 ln(1- 4/3p). Este un model simplu care presupune ca substitutiile sunt echiprobabile (α = β, unde α este probabilitatea de tranzitie, β este probabilitatea de transversie). In realitate, rata tranzitiilor este mai mare decat cea a transversiilor.
Modelul Kimura (K2P) tine cont de proportia dintre numarul de tranzitii (α) si transversii (β). Tranzitiile sunt mult mai numeroase decat transversiile. Formula corectiva propusa este: dxy = -1/2 ln(1- 2P -Q) + 1/4 ln (1- 2Q), unde P = proportia de tranzitii si Q = proportia de transversii.
UPGMA (Unweighted Pair-Group Method with Arithmetic means) este o metoda de clusterizare (cluster method) folositaa in bioinformatica pentru creare de arbori filogenetici printr-un algoritm de insumare secvential. Metoda se bazeaza pe regruparea secventelor celor mai apropiate. In matricea de distanta cele doua unitati taxonomice opertaionale avand distanta cea mai mica sunt adunate intr-un nou cluster compus, care va fi considerat in continuare ca o singura unitate operationala. Este creata o noua matrice, in care distantele celorlalte unitati taxonomice opertaionale ale clusterului sunt calculate. Procesul se repeta pana cand raman doar doua OTU. Acestea sunt insumate si radacina este amplasata la jumatatea distantei calculate dintre cele doua sume.
UPGMA este o metoda simpla, dezvoltata original pentru construirea fenogramelor taxonomice. Metoda se bazeaza pe asumarea unei rate constate a evolutiei sustinuta de ipoteza ceasului molecular si anume, distantele sunt ultrametrice si deci ele evolueaza cu o viteza constanta. Obtinerea de date ultrametrice este foarte improbabila, chiar daca rata substitutiei ar fi perfect constanta.
Metoda NJ (Neighbor Joing) este cea mai utilizata metoda de distanta, utilizand criteriul minimei evolutiei in constructia arborilor filogenetici. Ca si UPGMA, metoda NJ se bazeaza pe masurarea distantelor genetice pentru construirea unui arbore filogenetic, dar diferenta intre cele doua metode consta in faptul ca NJ tine cont de diferentele de viteza evolutiva intre diferitele ramuri ale arborelui filogenetic. Metoda NJ este mai eficienta decat celelate metode de distanta, utilizand un altgoritm polinominal ca si metodele de clusterizare. Metoda NJ nu utilizeaza ipoteza orologiului molecular, ci algoritmul utilizat in acest caz realizeza calculul lungimii bratelor arborelui, astfel incat distantele deduse ale arborelui sa corespunda cu cele mai apropiate distante masurate intre secvente. Aceasta modalitate de calcul este ilustrata si de denumirea metodei Neighbor Joing - "cel mai apropiat vecin". NJ genereaza arbori filogenetici fara radacina prin reunirea nodurilor care sunt apropiate unele de altele.
Metodele de distanta prezinta avantajul ca sunt metode rapide si permit analiza unui numar mare de date si testarea unui numar mare de ipoteze alternative. Dezavantajul acestor metode consta in pierderea unei parti de informatie la trecerea de al matricea bazata pe caractere la matrice bazata pe calculul distantelor. De asemenea, nu permit combinarea unor caractere diferite in aceeasi matrice (de ex. caractere morfologice si secvente ADN).
Metodele de caractere (cladistice)
Metoda MP ( Maximum Parcimony) este o metoda cladistica bazate pe parcimonie. Principiul parcimoniei postuleaza ca pentru un grup de specii, filogenia verosimila este cea care necesita cel mai mic numar de schimbari evolutive. Arborele filogenetic al speciilor este conceput implicand un numar minim de evenimente evolutive. Lungimea arborelui L este egala cu suma numarului de modificari I pentru fiecare situs k.
![]() ;
i=1
;
i=1
Etapele de analiza prin metoda MP sunt urmatoarele:
i. identificarea situsurilor informative;
ii. deducerea tuturor topologiilor arborilor posibili pentru secventele de date;
iii. calculul numarului minim de substitutii pentru fiecare situs informativ;
iv. calculul sumei de modificari pentru fiecare arbore;
v. alegerea topologiei arborelui care necesita cele mai putine modificari.
Metoda MP deduce arborele (sau arborii) cu cel mai mic numar de pasi necesari pentru a produce variatii ale secvenetei observate. MP prezinta o serie de avantaje: tine cont de tipurile de caractere, nu reduce informatia la un numar unic (o distanta), incerca sa furnizeze informatie despre secventele ancestrale si evalueaza arbori diferiti. Comparativ cu metodele de distanta este foarte lenta. De asemenea, prezinta dezavantajul de a nu utiliza decat partial informatia (situsuri informative), nu coreceteaza substitutiile multiple si nu calculeaza lungimea bratelor.
Metoda ML (Maximum Likelyhood) sau "metoda probabilitatii maxime" este o alternativa la metoda MP. ML este o metoda statistica utilizata pentru a deduce parametrii distributiei de probabilitate a unui anumit esantion. Din moment ce fiecare situs evolueaza independent, arborele este calculat separat pentru fiecare situs. Probabilitatae maxima este calculata pentru probabilitatea fiecarui rest dintr-un aliniament, pe baza unui model al procesului de substitutie. Prin metoda ML se incearca reconstruirea unei filogenii folosind un model explicit de evolutie. Metoda este cel mai eficienta cand este folosita pentru a testa sau a optimiza un arbore deja existent.
ML prezinta o serie de avantaje:
cea mai fiabila dintre celelate metode filogenetice, ceea care conduce la rezultatul cel mai apropiat de arborele evolutiv real.
comparativ cu parcimonia, ea este mai consistenta si mai putin sensibila la efectele atractiei bratelor lungi.
permite aplicarea a diferitelor modele evolutive (ex. modelul Kimura care tine cont de diferentele intre tranzitii si transversii) si de a estima lungimea bratelor in functie de scimbarea evolutie.
Principalul dezavantaj este ca cere o mare putere de calcul si mult timp, fiind cea mai lenta dintre toate metodele filogenetice.
Data fiind varietatea mare de metode care pot fi folosite in programele de filogenie este dificil pentru un biolog sa stabileasca care este cea mai buna metoda de analiza aunui set de date. Lucrarile publicate pe teme de filogenie propun folosirea unor algoritmi diferiti si a unor seturi de date diferite pentru obtinerea unor concluzii valabile. In unele cazuri, metode diferite actioneaza sinergic. De exemplu, metoda NJ produce un singur arbore care este utilizat pentru a valida un arbore construit prin metodele MP sau ML. Folosind metode alternative putem sa obtinem indicatii despre robustetea concluziilor rezultate. Principalul criteriu prin care se evalueaza acuratetea unui arbore filogenetic vizeaza consistenta, eficienta si robustetea. Evaluarea acuratetei se refera fie la o metoda filogenetica anume (de ex, UPGMA), fie la un aumit arbore.
Construirea si evaluarea arborilor filogenetici
Cel mai adesea termenul de filogenie este identificat cu un arbore filogenetic. Un arbore filogenetic este o reprezentare grafica a filogeniei unui grup de organisme, format din noduri si ramuri. Nodurile externe reprezinta unitati taxonomice operationale (OTU).
Exista mai multe tipuri de arbori (dendograme):
fenograma - o dendograma obtinuta prin metodele de distanta unde relatiile dintre taxoni exprima gradele de similitudine globala.
cladograma - o dendograma exprimand relatiile filogenetice dintre taxoni si construita plecand de la analiza cladistica.
filograma - o cladograma data de lungimea bratelor si proportionala cu numarul de schimbari evolutive.
Majoritatea metodelor filogenetice produc arbori fara radacina. Utilizarea unui outgroup (grup de comparatie)- grup ales din exteriorul grupului studiat duce la construirea unui arbore cu radacina.
In cazul arborilor elaborati pe baza metodei fenetice, lungimea ramurilor reprezinta distanta genetica dintre taxoni, iar pentru cei elaborati prin metoda cladistica (cladograme) acestea se raporteaza la evenimentele evolutive (caractere derivate) care au avut loc pentru fiecare linie.
Testarea fiabilitatii arborilor se face prin analiza " bootstrap". Analiza bootstrap este o abordare folosita de obicei pentru a masura robustetea topologiei unui arbore.
Bootstrap testeaza fiabilitatea ramurilor interne. Fiecare replicare boostrap produce o noua aliniere "artificiala"care este utilizata pentru a construi un arbore "artificial". Pentru fiecare ramura interna se calculeaza procentajul de arbori "artificiali" care contin aceasta ramificare. Consideram in general ca ramurile definite printr-o valoare de incarcare > 50% sunt fiabile. Sunt necesare 1000 de replicari de incarcare pentru ca aceasta metoda sa fie statistic valabila.
Analiza bootstrap poate fi aplicata arborilor filogenetici construiti prin metodele Neighbor Joining, Maximum Parsimony si UPGMA. Fiecare set de date bootstrap este analizat din punct de vedere filogenetic.
Construirea arborilor filogenetici pe baza secventelor pentru ARNr 16S, ARNr 12S si ARNt la salmonide a fost realizata prin metodele UPGMA, NJ, MP si ML cu programul PHYLIP (Phylogenetic inference package) v 3.68.
| Contact |- ia legatura cu noi -| | |
| Adauga document |- pune-ti documente online -| | |
| Termeni & conditii de utilizare |- politica de cookies si de confidentialitate -| | |
| Copyright © |- 2025 - Toate drepturile rezervate -| |
|
|
|||
|
|||
Analize pe aceeasi tema | |||
|
| |||
|
|||
|
|
|||








