
| Confectii | Diverse | Film televiziune | Fotografie | Pescuit |
Pescuit
|
|
Qdidactic » dezvoltare & ... » pescuit Construirea arborilor filogenetici pe baza secvențelor obținute |
Construirea arborilor filogenetici pe baza secvențelor obținute
Construirea arborilor filogenetici pe baza secvențelor obținute
Secventele pentru gena ARNr 16S au fost utilizate pentru construirea de arbori filogenetici prin diferite metode filogenetice. Stabilirea relatiilor filogenetice la speciile de salmonide provenind din diferite bazine hidrografice din Romania cu ajutorul metodelor de distanta (NJ - Neigbour Joining si UPGMA) s-a realizat utilizand diferite aplicatii ale programului Phylip 3.68. Pe baza matricei de distanta generate de DnaDist, utilizand ca model de substitutie Kimura2, au fost construiti arborii filogenetici prin metodele NJ (Figura 24) si UPGMA . Arborii rezultati au fost reprezentati sub forma unei filograme, respectiv arbore radial fara radacina.
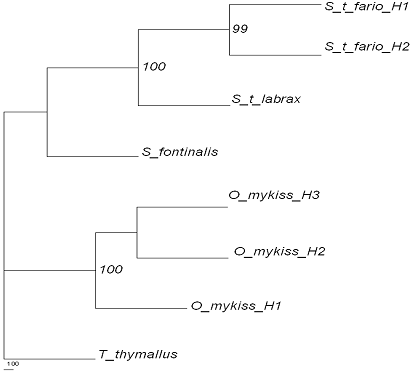
Figura 24: Filograma construita prin metoda NJ pe baza secventei genei mitocondriale ARNr 16S la speciile de salmonide din Romania. Model de substitutie Kimura2. Valoare de bootstrap: 1000 replicari.
Utilizand programul Phylip 3.68 au fost construiti arbori filogenetici pe baza metodelor de caractere ML (Maximum Likelihood) si MP (Maximum Parcimony) (Figurile 25-26), folosind ca valoare de bootstrap 1000 de replicari. Valorile de bootstrap obtinute prezinta valori >50, ceea ce indica o fiabilitate buna a arborilor obtinuti. Cea mai mica valoare de bootstrap in cazul metodei ML este de 55,8, iar in cazul metodei MP de 58,8.
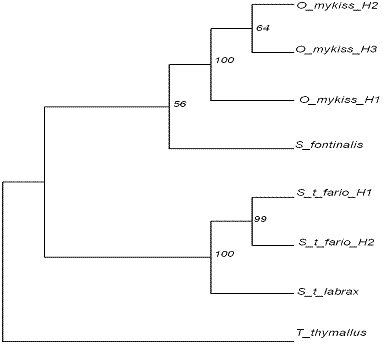
Figura 25: Relatiile filogenetice la speciile de salmonide din Romania pe baza secventei genei mitocondriale ARNr 16S obtinute prin metoda ML.
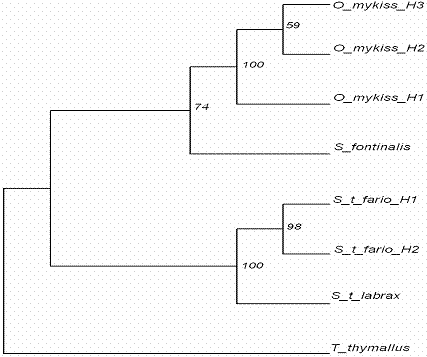
Figura 26: Relatiile filogenetice la speciile de salmonide din Romania pe baza secventei genei mitocondriale ARNr 16S obtinute prin metoda MP.
Desi, reprezentarile grafice ale arborilor filogenetici sunt diferite, relatiile filogenetice ilustrate sunt aceleasi.
La baza arborilor obtinuti se afla specia Thymallus thymallus. In acest caz datele moleculare reprezentate de secventa genei mitocondriale ARNr 16S confirmand datele de filogenie clasica care afirma ca subfamilia Thymallinae (reprezentant Thymallus thymallus) a aparut inaintea subfamiliei Salmoninae, care cuprinde genurile Salmo, Oncorhynchus, Hucho, Brachymystax si Salvelinus.
Salmo_trutta_fario_H1 si Salmo_trutta_fario_H2 ocupa pozitii echivalente, fiind vorba, de fapt, de haplotipuri mitocondriale ale aceleiasi specii. Aceasta specie prezinta cea mai mare apropiere filogenetica de Salmo trutta labrax, specie care apartine aceluiasi gen, Salmo. Reprezentantii genului Salmo formeaza o clada distincta, inrudita cu clusterul format de Salvelinus/Oncorhynchus.
Desi, datele de filogenie clasica sustin ca genurile Salmo si Onchorhynchus sunt direct inrudite, anumite date de filogenie moleculara afirma ca intre genurile Salvelinus si Oncorhynchus exista o apropiere filogenetica mai mare. Oakley & Phillips (1999), au stabilit relatiile filogenetice la 18 specii de salmonide utilizand un marker molecular nuclear (intronii genelor GH1 si GH2). Relatiile de filogenie clasica intre Oncorhynchus si Salmo au fost sustinute de analiza MP, dar nu si de ML.
In concluzie, relatiile filogenetice intre Salmo si Oncorhynchus ca taxoni direct inruditi sunt controversate, aceiasi autori propunand ipoteza ca Oncorhynchus si Salvelinus, sunt mai apropiate din punct de vedere filogenetic decat Oncorhynchus si Salmo. Crespi et al., (2003) au realizat un studiu complex de filogenie la 30 de specii de salmonide, utilizand ca markeri filogenetici 16 gene mitocondriale si 9 gene nucleare. Printre concluziile acestui studiu se numara si faptul ca (a) Salmo reprezinta un grup inrudit cu grupul (Oncorhynchus, Salvelnius), (b) Salvelinus este taxonul cel mai apropiat din punct de vedere filogenetic de Oncorhynchus. Concluziile anterioare au fost bine sustinute de valorile de bootstrap obtinute si de analiza de tip bayesian. Inrudirea intre Salvelinus si Oncorhynchus a fost sustinuta de analiza genelor GH1C, VIT si ND3 si de datele nucleare combinate.
In cazul analizei noastre bazate pe secventa ARNr 16S, interpunerea speciei Salvelinus fontinalis intre reprezentantii genului Salmo-Salmo trutta fario si Salmo trutta labrax, si Oncorhynchus mykiss este sustinuta atat de analiza de tip MP, cat si de analiza de tip ML. Haplotipurile mitocondriale ale Oncorhynchus mykiss formeaza o clada separata, O_mykiss_H2 si O_mykiss_H3 fiind mai apropiate intre ele decat cu O_mykiss_H1. Acest fapt confirma faptul ca exista mai multe varietati ale acestei specii in pastravariile din Romania.
In cazul construirii arborilor de distanta prin metoda NJ, apar diferente in ceea ce priveste pozitia ocupata de Salvelinus fontinalis, in functie de modelul de substitutie ales. In cazul in care alegem ca model de substitutie LogDet, topologia arborelui obtinut prin NJ este similara cu cea a arborilor obtinuti prin MP si ML (Figura 27). Distanta de tip LogDet calculeaza frecventa cu care apar diferentele dintre cele patru nucleotide intre liniile analizate. Se opteaza pentru parametrul LogDet cand frecventa bazelor in cazul speciilor analizate nu este constanta.
In cazul in care am optat in construirea matricei de distanta pentru celelalte modele de substitutie oferite de aplicatia DnaDist (F84, Jukes-Cantor, Kimura2), topologia arborilor rezultati este identica si prezinta specia Salvelinus fontinalis, formand un grup monofiletic cu speciile genului Salmo, distinct de grupul format de specia Oncorhynchus mykiss.
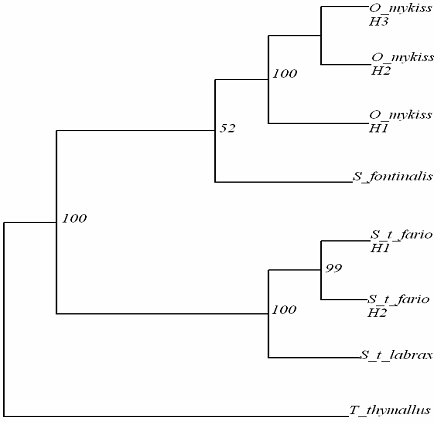
Figura 27: Relatiile filogenetice la speciile de salmonide din Romania pe baza secventei genei mitocondriale ARNr 16S obtinute prin metoda NJ-model de substitutie LogDet.
In cazul genei mitocondriale, ARNr 12S, la cele 5 specii de salmonide din Romania au fost analizate 861 de situsuri nucleotidice, avand compozitia in baze azotate prezentata in tabelul 4.
Tabel 4: Compozitia in nucleotide a fragmentului amplificat pentru gena ARNr 12S la speciile de salmonide din Romania.
|
Secventa |
Lungime (pb)
|
G+C |
A+T |
Compozitie nucleotide |
||||
|
S_t_fario |
860 |
51,16% |
48,84% |
A 252 (29,30%) C 247 (28,72%) G 193 (22,44%) T 168 (19,53%) |
||||
|
S_t_labrax |
860 |
50,93% |
49,07% |
A 254 (29,53%) C 246 (28,60%) G 192 (22,33%) T 168 (19,53%) |
||||
|
O_mykiss H1 |
860 |
50,81% |
49,19% |
A 256 (29,77%) C 245 (28,49%) G 192 (22,33%) T 167 (19,42%) |
|
O_mykiss H2 |
860 |
50,93% |
49,07% |
A 255 (29,65%) C 245 (28,49%) G 193 (22,44%) T 167 (19,42%) |
|
O_mykiss H3 |
861 |
50,75% |
49,25% |
A 258 (29,97%) C 245 (28,46%) G 192 (22,30%) T 166 (19,28%) |
|
S_fontinalis |
861 |
50,47% |
49,53% |
A 256 ( 29,77%) C 241 (28,02%) G 193 (22,44%) T 170 (19,77%) |
|
T_thymallus |
859 |
50,76% |
49,24% |
A 250 (29,10%) C 240 (27,94%) G 196 (22,82%) T 173 (20,14%) |
Secventele nucleotidice obtinute pentru un fragment din gena mitocondriala ARNr 12S la salmonidele din Romania au fost aliniate cu programul ClustalW. Din cele 861 de situsuri nucleotidice, 833 sunt situsuri identice.
Dintre polimorfismele interspecifice inregistrate la nivelul seventei genei ARNr 12S, 20 sunt tranzitii si 7 sunt transversii, raportul Ts/Tv fiind de 2,85.
Dintre cele 861 situsuri nucleotidice analizate 32 de situsuri sunt informative din punct de vedere parcimonic.
Procentul de situsuri nucleotidice variabile este mult mai mare in cazul secventelor pentru ARNr 16S (20,1%) comparativ cu procentul situsurilor variabile in cazul secventelor pentru ARNr 12S (3,3%). O diferenta semnificativa se pastreaza si pentru situsurile informative din punct de vedere parcimonic ( Pi) la cele doua tipuri de fragmente analizate- 9,38% situsuri Pi in cazul ARNr 16S vs. 3,7% situsuri Pi in cazul ARNr 12S.
Secventele pentru ARNr 12S au fost utilizate pentru construirea de arbori filogenetici prin diferite metode filogenetice. Pe baza matricei de distanta generate de DnaDist au fost construiti arborii filogenetici prin metodele NJ (Figura 28) si UPGMA . Indiferent de modelul de substitutie ales (F84, K2P, JC, LogDet) pentru generarea matricei de distanta, au fost obtinuti arbori filogenetici similari, deci in cazul secventelor obtinute pentru ARNr 12S, modelul de substitutie ales nu influenteaza topologia arborilor. Arborii rezultati prin metodele de distanta au fost reprezentati sub forma unei filograme, respectiv arbore radial fara radacina.
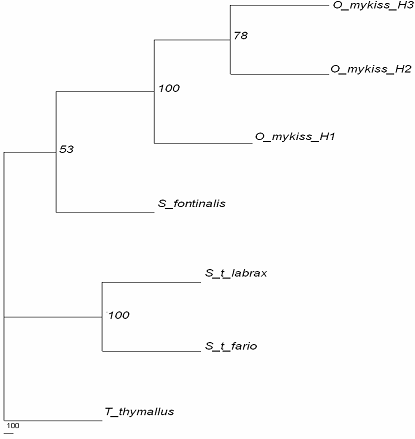
Figura 28: Filograma construita prin metoda NJ pe baza secventei genei mitocondriale ARNr 12S la speciile de salmonide din Romania. Model de substitutie: Kimura 2P. Valoare de bootstrap: 1000 de replicari.
Cu ajutorul programului Phylip 3.68 au fost construiti arbori filogenetici pe baza metodelor de caractere ML si MP (Figurile 29-30), utilizand ca valoare de bootstrap 1000 de replicari. Valorile de bootstrap obtinute prezinta valori >50, ceea ce indica o fiabilitate buna a arborilor obtinuti.
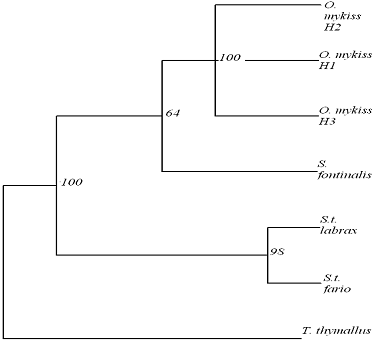
Figura 29: Relatiile filogenetice la speciile de salmonide din Romania pe baza secventei genei mitocondriale ARNr 12S obtinute prin metoda MP.
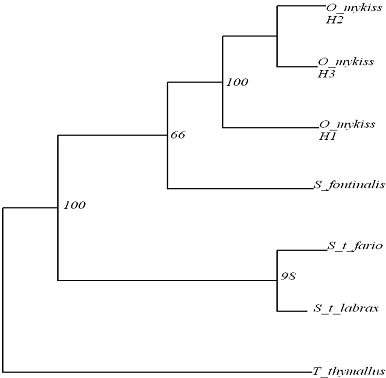
Figura 30: Relatiile filogenetice la speciile de salmonide din Romania pe baza secventei genei mitocondriale ARNr 12S obtinute prin metoda ML.
Arborii rezultati folosind ca marker filogenetic un fragment din gena mitocondriala pentru ARNr 12S la salmonidele din Romania ilustreaza relatiile filogenetice la aceste specii. Similar anailzei filogenetice bazate pe ARNr 16S, specia Thymallus thymallus ocupa o pozitie bazala, ca ancestor al celorlate specii de salmonide. Speciile Salmo trutta fario si Salmo trutta labrax formeaza o clada distincta de cea reprezentata de Salvelinus fontinalis/ Oncorhynchus mykiss. Salvelinus fontinalis apare ca fiind mai apropiata din punct de vedere filogenetic de genul Oncorhynchus decat de genul Salmo.
Pentru fragmentul din gena ARNtPro/Thr la cele 5 specii au fost analizate 149 de situsuri nucleotidice, avand compozitia in baze azotate prezentata in tabelul 5.
Tabel 5: Compozitia in nucleotide a fragmentului ARNt de la speciile de salmonide din Romania.
|
Secventa |
Lungime (pb) |
G+C |
A+T |
Compozitie nucleotide |
|
S_t_fario |
149 |
52,35% |
47,65% |
A 40 (26.85%) C 41 (27.52%) G 37 (24.83%) T 31 (20.81%) |
|
S_t_labrax |
149 |
52,35% |
47,65% |
A 40 (26.85%) C 41 (27.52%) G 37 (24.83%) T 31 (20.81%) |
|
O_mykiss |
149 |
54,36% |
45,64% |
A 39 (26.17%) C 45 (30.20%) G 36 (24.16%) T 29 (19.46%) |
|
S_fontinalis |
149 |
53,02% |
46,98% |
A 39 (26.17%) C 42 (28.19%) G 37 (24.83%) T 31 (20.81%) |
|
T_thymallus |
149 |
53,02% |
46,98% |
A 41 (27.52%) C 42 (28.19%) G 37 (24.83%) T 29 (19.46%) |
Secventele nucleotidice obtinute pentru un fragment din gena mitocondriala ARNtPro/Thr au fost aliniate cu programul ClustalW. Din cele 149 de situsuri nucleotidice, 141 sunt situsuri identice. Dintre polimorfismele interspecifice inregistrate la nivelul seventei genei ARNt, 6 sunt tranzitii si 2 sunt transversii, raportul Ts/ Tv fiind 3.
Dintre cele 149 de situsuri nucleotidice analizate, 8 sunt informative din punct de vedere parcimonic.
Utilizand ca marker filogenetic gena mitocondriala pentru ARNtPro/Thr, au fost construiti arbori filogenetici prin metodele de distanta NJ si UPGMA si prin metodele de caractere ML si MP.
Pe baza matricei de distanta generate de DnaDist (program Phylip 3.68) au fost construiti arborii filogenetici prin metodele NJ (Figura 31) si UPGMA. Indiferent de modelul de substitutie ales (F84, K2P, JC, LogDet) pentru generarea matricei de distanta, au fost obtinuti arbori filogenetici similari, deci topolgia arborilor rezultati folosind ca marker filogenetic ARNtPro/Thr nu este influentata de modelul de substitutie ales.
Arborii rezultati prin metodele de distanta au fost reprezentati sub forma unei filograme, respectiv arbore radial fara radacina.
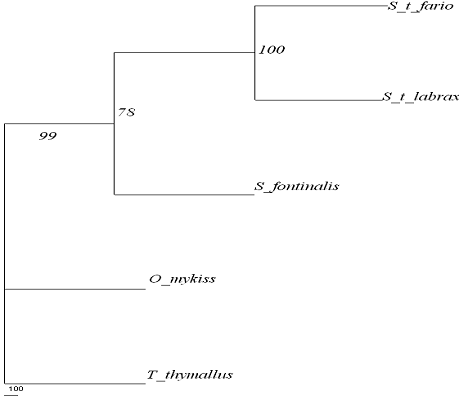
Figura 31: Filograma construita prin metoda NJ pe baza secventei genei mitocondriale ARNtPro/Thr la speciile de salmonide din Romania. Model de substitutie: Kimura 2P. Valoare de bootstrap: 1000 de replicari.
Cu ajutorul programului Phylip 3.68 au fost construiti arbori filogenetici pe baza metodelor de caractere MP si ML (Figurile 32-33), utilizand ca valoare de bootstrap 1000 de replicari.
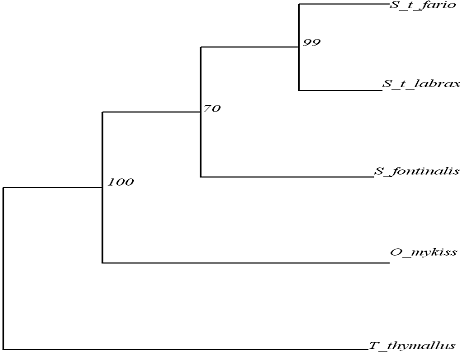
Figura 32: Cladograma rezultata prin metoda MP pe baza secventei genei mitocondriale ARNtPro/Thr la speciile de salmonide din Romania.
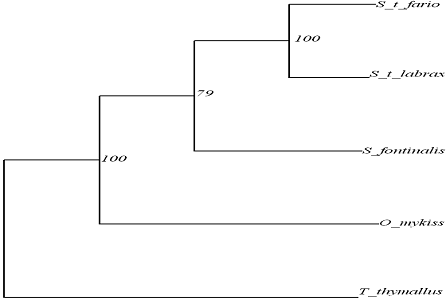
Figura 33: Cladograma rezultata prin metoda ML pe baza secventei genei mitocondriale ARNtPro/Thr la speciile de salmonide din Romania.
Indiferent de tipul de metoda folosita, arborii rezultati indica aceleasi relatii filogenetice la speciile de salmonide din tara noastra. Diferit, de relatiile filogenetice stabilite utilizand ca markeri filogenetici genele mitocondriale ARNr 16S si ARNr 12S, in cazul ARNt specia Salvelinus fontinalis, formeaza un grup monofiletic cu speciile Salmo trutta fario si Salmo trutta labrax, si nu cu Oncorhyncus mykiss, asa cum s-a stabilit anterior.
Lungimea redusa a fragmentului ARNtPro/Thr de 149 de nucleotide comparativ cu 864 si 861 de nucleotide pentru ARNr 16S si ARNr 12S, poate constitui un impediment in analiza filogenetica moleculara si astfel se explica incongruenta datelor obtinute.
Datele mitocondriale pentru cele trei fragmente genice ARNr 16S, ARNr 12S si ARNt (in total 1873 de situsri nucleotidice) au fost analizate impreuna pentru stabilirea relatiilor filogenetice la speciile de salmonide din Romania si au fost construiti arbori filogenetici prin metodele NJ, UPGMA, MP si ML (Figurile 34-37).
Arborii rezultati prin metodele de distanta au prezentat aceeasi topologie indiferent de modelul de substitutie ales pentru construirea matricei de distanta, deci putem afirma ca in cazul datelor mitocondriale combinate modelul de substitutie ales nu influenteaza semnificativ relatiile filogenetice rezultate. Indiferent de metoda aleasa, arborii au ilustrat relatii filogenetice similare si au prezentat o fiabilitate buna, cu valori de bootstrap >50.
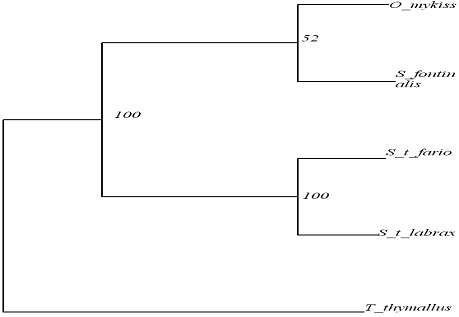
Figura 34: Cladograma rezultata prin metoda NJ utilizand datele mitocondriale reunite (ARNr 16S, ARNr 12S, ARNtPro/Thr) la speciile de salmonide din Romania. Model de substitutie K2P. Valoare de bootstrap - 1000 replicari.
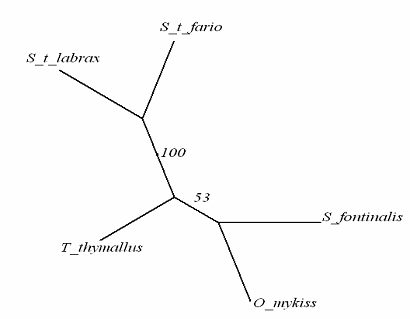
Figura 35: Arbore filogenetic de tip radial, fara radacina, construit prin metoda UPGMA pe baza secventei genei mitocondriale ARNtPro/Thr la speciile de salmonide din Romania.
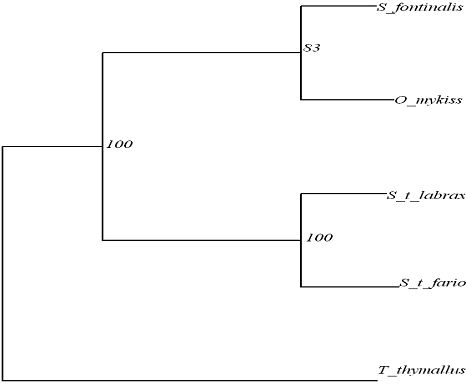
Figura 36: Cladograma rezultata prin metoda MP utilizand datele mitocondriale reunite (ARNr 16S, ARNr 12S, ARNtPro/Thr) la speciile de salmonide din Romania.
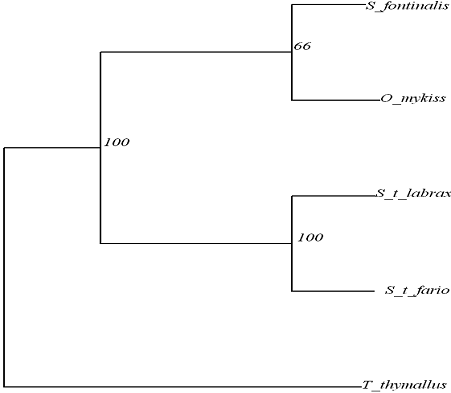
Figura 37: Cladograma rezultata prin metoda ML utilizand datele mitocondriale reunite (ARNr 16S, ARNr 12S, ARNtPro/Thr) la speciile de salmonide din Romania.
Relatiile filogenetice existente intre speciile de salmonide din Romania stabilite pe baza datelor mitocondriale reunite (ARNr 16S, ARNr 12S, ARNtPro/Thr) arata ca:
Thymallus thymallus reprezinta ancestorul celorlalte specii de salmonide analizate, fiind specia cea mai primitiva si care a sta la originea formarii celorlate specii de salmonide.
Speciile autohtone apartinand genului Salmo - Salmo trutta fario si Salmo trutta labrax sunt strans inrudite formeaza o clada distincta de grupul Salvelinus fontinalis/ Oncorhynchus mykiss.
Salvelinus fontinalis este cea mai apropiata din punct de vedere filogenetic de Oncorhynchus mykiss, cu care formeaza un grup monofiletic, contrazicand astfel ipoteza filogeneiei clasice ca Salmo si Oncorhynchus sunt specii "surori".
Pentru stabilirea unor relatii filogenetice mai complexe, markerii filogenetici ARNr 16S si ARNr 12S au fost analizati pe langa speciile de salmonide din Romania si la alte specii de salmonide, pentru acestea utilizand secvente nucleotidice din GenBank.
| Contact |- ia legatura cu noi -| | |
| Adauga document |- pune-ti documente online -| | |
| Termeni & conditii de utilizare |- politica de cookies si de confidentialitate -| | |
| Copyright © |- 2025 - Toate drepturile rezervate -| |
|
|
|||
|
|||
Referate pe aceeasi tema | |||
|
| |||
|
|||
|
|
|||








